Investigadores de la Universidad Politécnica de Madrid han creado una herramienta que permite homogeneizar y compartir datos clínicos y genómicos entre instituciones sanitarias europeas.
El Grupo de Informática Biomédica de la Universidad Politécnica de Madrid ha desarrollado un software llamado “Semantic Interoperability Layer” –SIL– que utiliza terminologías estándar como un vehículo para abordar los dos desafíos principales de la interoperabilidad multicéntrica: armonizar las heterogeneidades de diferentes fuentes de datos e integrar los datos ómicos y clínicos para mejorar la prevención, el diagnóstico y las terapias de distintas enfermedades, entre ellas, el cáncer de mama.

El Grupo de Informática Biomédica se creó oficialmente en 1993 y forma parte del Departamento de Inteligencia Artificial de la ETSI de Ingenieros informáticos. El GIB está formado por más de 20 investigadores y un gran número de colaboradores. / Fotografía: UPM
Facilitar el intercambio de datos
La introducción de datos ómicos (aquellos derivados de las tecnologías ómicas, como la genómica, la transcriptómica, la proteómica, etc.) como parte de las pruebas utilizadas en la práctica clínica actual, ha creado una gran estratificación de los pacientes basada en sus perfiles genéticos. Hace años se podían conseguir dentro de un mismo hospital o centro de salud los pacientes necesarios para llevar a cabo un ensayo clínico, sin embargo hoy en día la obtención de una cohorte mínima de pacientes para un estudio de este tipo obliga a involucrar frecuentemente a distintos hospitales de diferentes regiones o países.
En este contexto, el intercambio de datos entre distintos centros es complicado, no sólo por aspectos legales, sino también por aspectos técnicos. Los datos requeridos para los estudios, se almacenan en cada hospital, e incluso en cada departamento dentro de un hospital, en sistemas de información heterogéneos que siguen diferentes formatos y están codificados en distintas terminologías médicas e idiomas.

La nueva herramienta permite homogeneizar y compartir datos clínicos y genómicos. / Fotografía: Pixabay
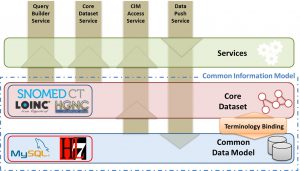
El SIL “se compone de cuatro partes principales: un modelo de datos común (Common Data Model) capaz de enlazarse de una manera estándar con los sistemas de información de los hospitales; un vocabulario común (Core Dataset) para la codificación de la información y datos provenientes de los distintos hospitales; un enlace automático (Terminology Binding) entre el modelo de datos común y el vocabulario; y una serie de servicios para acceder y administrar la información almacenada”, explica uno de los investigadores a cargo.
Compartir para lograr una mayor eficiencia
Las principales novedades de esta capa de interoperabilidad es que los datos no son sólo traducidos al vocabulario común, sino que son además normalizados. Adicionalmente, la capa de interoperabilidad es capaz de almacenar datos de pruebas genéticas en el mismo almacén de datos que los datos clínicos, de manera que toda la información pueda ser guardada y consultada de una forma homogénea.
Esta capa de interoperabilidad ha sido probada con datos reales de hospitales de España, Alemania, Bélgica, Holanda y Reino Unido dentro de la colaboración en varios proyectos nacionales e internacionales. El trabajo, llevado a cabo por los miembros del Grupo de Informática Biomédica de la Universidad Politécnica de Madrid en la ETSI Informáticos, ha sido publicado en varias revistas científicas internacionales.
Fuente: UPM
Boletín original: http://www.upm.es/UPM/SalaPrensa/Noticias_de_investigacion?id=3ffd6754b05af510VgnVCM10000009c7648a____&fmt=detail&prefmt=articulo


Deja una respuesta