Biólogos genetistas e informáticos de la Universidad del País Vasco –UPV/EHU-, Euskal Herria, han aunado esfuerzos para crear un software, WREGEX 2.0, que analiza en las proteínas mutaciones, posibles inductoras de enfermedades como el cáncer. Este programa es una aplicación bioinformática libre, fácil, versátil y, sobre todo, rápida que puede llegar a analizar y combinar la información de 40.000 proteínas en un minuto.
Lograr leer los motivos funcionales

El equipo de investigadores. De izquierda a derecha: José Antonio Rodríguez, Gorka Prieto y Asier Fullaondo / Foto: UPV/EHU
Las proteínas están formadas por cadenas de aminoácidos, y en cada proteína se pueden distinguir secuencias de aminoácidos cortas con una función discreta, llamadas motivos funcionales. En ocasiones estos motivos ya están descritos, y en otras están por definir. Cuando un motivo funcional aparece modificado, es posible que esa mutación influya en el desarrollo de una enfermedad como el cáncer. Verificar los posibles cambios en una proteína es uno de los primeros pasos parar llevar adelante una investigación sobre su función.
Bioinformática al servicio de la salud
Por esta razón, el equipo de investigadores empezó a trabajar en la herramienta bioinformática. En un principio, desarrolló un software –WREGEX– que sirve para predecir y buscar de manera automática ‘motivos funcionales’, es decir los pequeños grupos de aminoácidos que desarrollan tareas específicas de una proteína.
Después, dio un paso adicional y combinó la información sobre la secuencia de todas las proteínas humanas conocidas con el catálogo COSMIC que recoge las mutaciones relacionadas con el cáncer. Y así, apareció WREGEX 2.0, nueva versión que permite comparar una proteína normal y la misma mutada para, de esta forma, poder predecir ‘motivos funcionales’ que hayan sido modificados y puedan tener una relación con el cáncer.
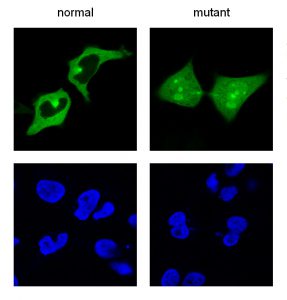
Una mutación del aminoácido 144 de la proteína UPSP21 (que cuenta con más de 500 aminoácidos) hace que esta proteína se acumule en el núcleo de una célula, en lugar de estar fuera. La imagen, obtenida mediante microscopía fluorescente, muestra la proteína en verde y el núcleo de la célula en azul / Foto: UPV/EHU
La herramienta combina tres tipos de informaciones: las secuencias de proteínas, los motivos funcionales y las mutaciones del cáncer. «Una de las principales características de WREGEX 2.0 es que puede estudiar simultáneamente proteomas muy complejos, con muchísimas proteínas, y combinar información, en el caso del ensayo, con mutaciones de cáncer; pero la puerta está abierta a utilizar otras bases de datos que contengan información sobre otro tipo de mutaciones. La ventaja, además, es que en un minuto se pueden analizar 40.000 proteínas, mientras que con otros programas el análisis de una proteína llevaba varios minutos«, explicaba uno de los científicos involucrado en el proyecto. Por lo tanto, mediante este software, se puede predecir que la alteración en una proteína puede influir en el desarrollo de la enfermedad, no sólo cáncer.
WREGEX 2.0: conocimiento libre disponible en la red
Cabe señalar que WREGEX 2.0 está disponible para la comunidad científica en el servidor de la UPV/EHU (http://ehubio.ehu.eus/wregex ). Hasta ahora, ya hay trece trabajos de investigación que han utilizado esta herramienta informática. Y al servidor han entrado investigadores de China, Japón, Corea, Alemania y Estados Unidos.
Fuente: UPV

Deja una respuesta